Vous êtes ici
Les technologies single cell révolutionnent la recherche en biologie
À l’Institut Curie, l’échantillon d’une tumeur circule d’étage en étage. D’abord prélevé par un médecin sur une patiente au sous-sol, l’échantillon monte au service de pathologie pour être préparé. Il est ensuite analysé cellule par cellule grâce à une technologie révolutionnaire baptisée « single cell » (« cellule unique ») et finit par remonter encore d’un étage pour être séquencé. Enfin, des bio-informaticiens nettoient le jeu de données produit par le séquenceur, et lui appliquent des méthodes statistiques pour l’étudier. Le but : comprendre pourquoi une tumeur émerge et résiste au traitement. En quelques mois, l’échantillon vivant s’est transformé en données. « Les méthodes de single cell ont accéléré la biologie 2.0, nos travaux reposent sur un ballet pluridisciplinaire, qui va du médecin au biologiste, à l’analyste de données et au statisticien. C’est parce que toutes ces personnes s’y mettent main dans la main que tout fonctionne », décrit Céline Vallot, directrice de recherche CNRS à l’Institut Curie1, spécialiste des mécanismes d’épigénétique du cancer du sein.
Depuis 2017, Céline Vallot et son équipe utilisent et développent des méthodes de single cell pour la recherche sur le cancer. Le single cell est une innovation qui utilise un procédé appelé « microfluidique » : les cellules passent une à une dans des micro-canaux pour être encapsulées dans une micro-gouttelette d’huile contenant des réactifs. Au moment de l’encapsulage, le matériel génétique de la cellule est marqué par un code-barres génétique qui permet d’identifier la cellule. Grâce à cette technologie, la chercheuse se place à l’échelle de la cellule « unique », et essaie de comprendre l’hétérogénéité cellulaire au sein d’une même tumeur. « Avant le single cell, on faisait des expériences sur un groupe de millions de cellules, on étudiait l’expression moyenne des gènes », explique Céline Vallot.
Pourtant, la chercheuse précise que l’échelle de la cellule unique n’est pas nouvelle en biologie. « Dans l’étude des protéines, on utilisait des méthodes de microscopie. Ce qui est novateur avec le single cell, c’est le côté systématique, on peut étudier beaucoup de cellules et avoir une information relativement exhaustive », ajoute-t-elle. Cette nouvelle technique porte de nombreux espoirs pour la recherche sur le cancer et en médecine, avec une perspective majeure : créer une médecine de précision où chaque patient aurait un traitement spécifique.
Des approches multiples
La première publication scientifique2 où des ARN sont étudiés avec la méthode du single cell date de 2009. En 2015, la technologie s’accélère et à partir de 2017, la plupart des laboratoires de biologie étudiant le cancer en France se dotent de cette technique. Au Centre de recherche en cancérologie et immunologie intégrée Nantes-Angers3, la machine est arrivée en 2018. Pas plus grande qu’un mini lave-vaisselle, elle a ouvert un monde des possibles pour l’équipe de Stéphane Minvielle, directeur de recherche CNRS et co-responsable de l’équipe Integrative Cancer Genomics. Depuis vingt ans, l’équipe installée au cœur du CHU de Nantes étudie le myélome multiple, un cancer de la moelle osseuse. Leurs travaux se concentrent sur l’activité des cellules, par le biais de leurs ARN messagers : c’est l’approche transcriptomique, ou l’étude et l’analyse de la transcription de l’ADN en ARN messagers.
La combinaison du single cell et de la transcriptomique est baptisée « single cell RNA-seq ». « Grâce à cette technique, on peut différencier les sous-types de cellules du système immunitaire alors qu’avant, on ne voyait qu’un bloc, on accède à des réponses complexes sur comment la cellule tumorale modifie son environnement », explique Stéphane Minvielle. La technique du single cell peut être combinée à d’autres protocoles expérimentaux. Céline Vallot étudie l’épigénétique, c’est-à-dire l’activité de la cellule à travers les mécanismes de régulation de l’expression des gènes. « Nous appliquons la méthode du single cell à l’épigénétique, ce que peu de laboratoires dans le monde font », précise-t-elle. L'association de ces deux techniques est quant à elle baptisée « single cell ChIP seq ».



À l’Institut de génomique fonctionnelle de Lyon4, Yad Ghavi-Helm travaille sur l'embryon de la mouche du vinaigre, Drosophila melanogaster. L’objectif est d’utiliser la drosophile comme modèle pour comprendre les interactions entre les régions régulatrices du génome, aussi appelées enhancers, et les gènes, « car des mutations dans la séquence des enhancers peuvent affecter l'expression des gènes qu'ils régulent et causer des maladies telles que des cancers », détaille la chercheuse. Comme à Nantes, Yad Ghavi-Helm associe le single cell RNA-seq à la transcriptomique spatiale, une technologie qui combine le profilage à haut débit de l’expression des gènes avec l’information de la localisation spatiale des cellules dans leurs tissus ou organes d’origine. « Jusqu'à présent, seules les méthodes d'imagerie permettaient d'étudier l'expression des gènes de cette façon, mais cette technique restait limitée à une poignée de gènes par expérience. Avec la transcriptomique spatiale on peut analyser des centaines, voire des milliers de gènes en parallèle », développe-t-elle.
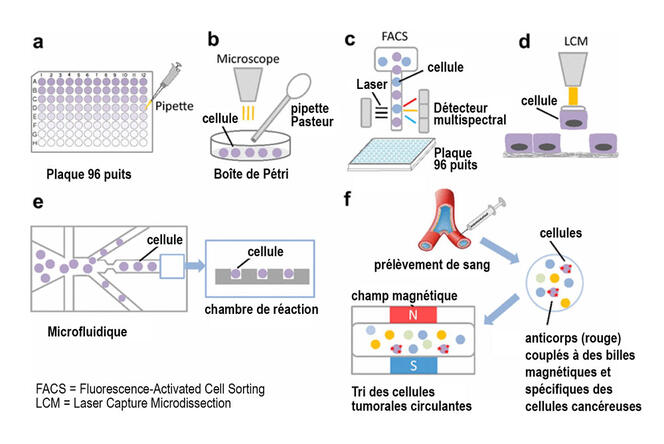

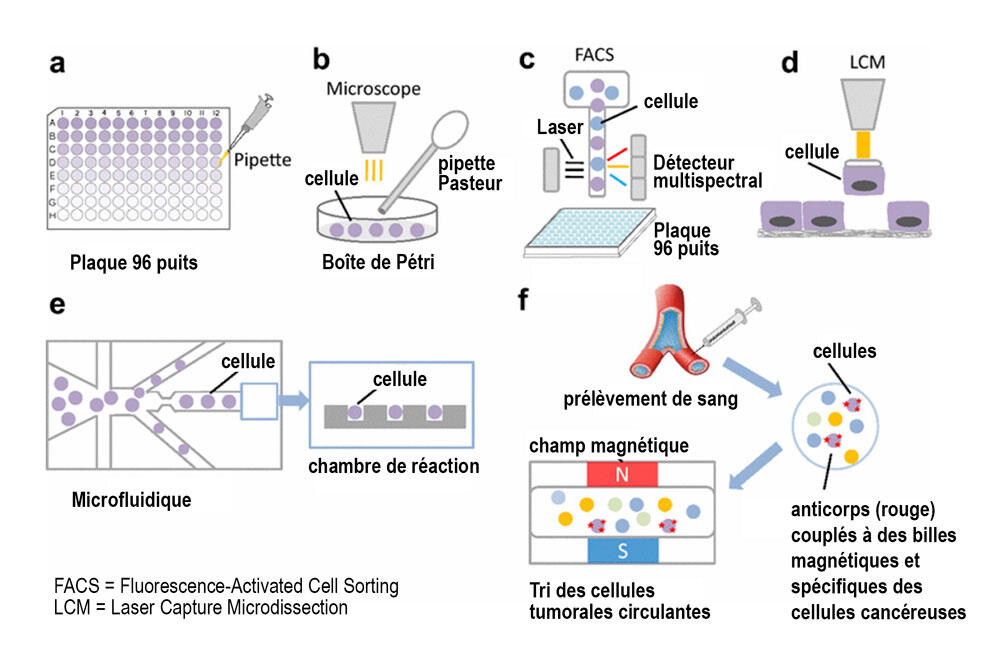
D’autre part, les méthodes de single cell approfondissent aussi la compréhension du rôle des protéines dans les cellules, leur modification et leur dégradation. La protéomique désigne la science qui étudie les protéomes, c'est-à-dire l'ensemble des protéines d’une cellule. Avant le single cell, avoir accès aux protéines étaient un défi majeur : très sensibles, ne pouvant être amplifiées, les protéines doivent être analysées à très petite échelle ce qui rend les méthodes classiques comme la spectrométrie de masse peu précises et performantes. En se plaçant à l’échelle de la cellule unique, le single cell appliqué à la protéomique (SCP) aborde des questions jusqu’ici inexplorées par les anciennes méthodes. Le single cell avance si vite que ses limites techniques sont en passe d’être franchies, mais son coût d’utilisation demeure un frein majeur pour les laboratoires.
Mettre le vivant en équations
« Avec l’arrivée du single cell, on est passé d’un tableau Excel de 20 000 lignes à des serveurs de calculs », affirme Stéphane Minvielle. Le single cell a entraîné une massification de la production des données, « ceux qui produisent les données ne peuvent plus les interpréter seuls, les biologistes ont besoin de s’appuyer sur des ingénieurs et aussi sur une recherche méthodologique en statistique et en intelligence artificielle », éclaire Franck Picard, directeur de recherche CNRS au Laboratoire de biologie et modélisation de la cellule5, spécialiste de statistique et de machine learning (apprentissage automatique), au cœur de la révolution du single cell. Céline Vallot abonde dans ce sens : « J’ai l’impression qu’on utilise 5 % de ce que les données nous disent. Si on était aussi forts que ChatGPT à analyser nos données, ce serait bien. Comment se doter des moyens et des machines pour tirer un maximum d’informations de ces données ? ». Le single cell implique une forte interdisciplinarité, il crée des ponts entre les biologistes et ceux qui font de la recherche en méthodes d'analyse de données ; par conséquent, attirer des mathématiciens vers la biologie devient une nécessité.
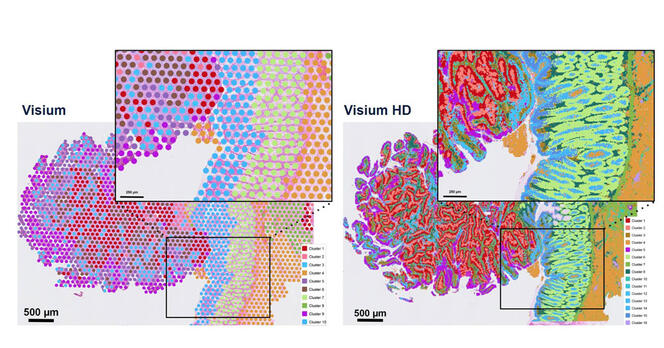

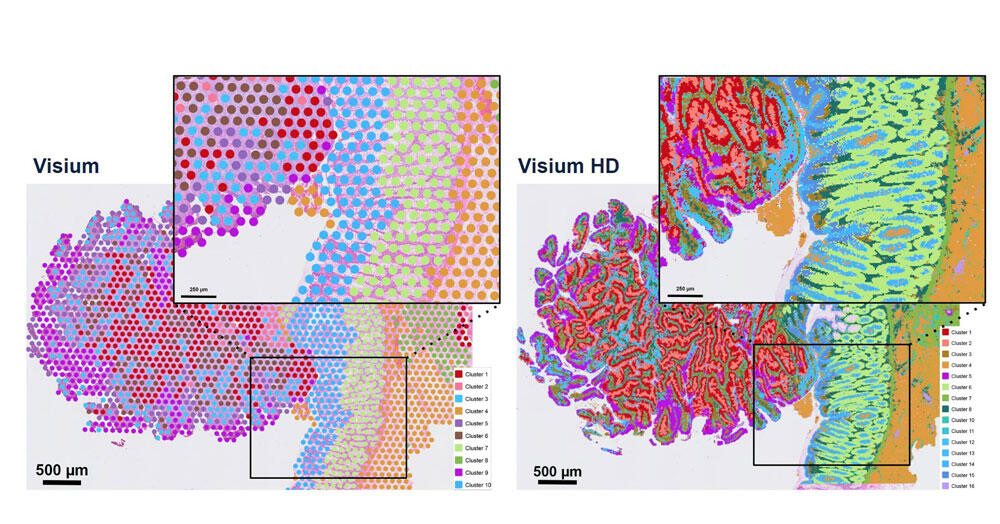
« Le défi pour la recherche méthodologique est de mettre en équation le vivant », résume Stéphane Minvielle. « En France, notre avantage est que la formation en maths et en machine learning est excellente », ajoute Franck Picard. Mais en intelligence artificielle (IA), la compétitivité est grande et ces profils ont tendance à être recrutés dans le privé où les salaires sont plus importants que ceux de la recherche publique. Pour y remédier, Franck Picard et Céline Vallot misent sur la formation doctorale. « Il faut valoriser et encourager les thèses pour construire ces profils hybrides sur la durée », affirme Franck Picard, avec pour idéal de faire aussi émerger plus d’instituts interdisciplinaires. Dans cette dynamique, depuis 2023, le single cell est mis en avant dans le Programme et équipement prioritaire de recherche Santé numérique6, copiloté par l’Inserm et Inria. Le consortium réunit des experts en analyse de données en single cell. « Le but, explique Franck Picard qui coordonne un des projets du programme pour le CNRS7, est de développer de nouvelles méthodologies en IA pour l'exploitation des données du single cell. L'objectif à terme est de faire un lien avec la médecine de précision et surtout, de faire émerger une communauté française spécialiste de ce domaine. » La révolution n’en est qu’à ses prémisses. ♦
- 1. Dynamique de l'information génétique : bases fondamentales et cancer (DIG-CANCER), unité CNRS/Institut Curie/Sorbonne Université.
- 2. Tang, F., Barbacioru, C., Wang, Y. et al. mRNA-Seq whole-transcriptome analysis of a single cell. Nat Methods 6, 377–382 (2009). https://doi.org/10.1038/nmeth.1315
- 3. Unité CNRS/Inserm/Nantes Université/Université d'Angers.
- 4. Unité CNRS/ENS Lyon.
- 5. Unité CNRS/ENS Lyon.
- 6. https://pepr-santenum.fr/
- 7. IA multi-échelle pour une médecine de précision en cellules uniques (AI4scMED). https://pepr-santenum.fr/2023/11/06/ai4scmed/